空色天絵
728 字
4 分钟
使用 LocalColabFold 本地部署 AlphaFold 2

为什么要本地部署 AlphaFold?
- 数据隐私:药企和科研机构的序列数据高度敏感,必须通过“物理隔离”保护,避免上传至云端。
- 高通量需求: 突破 Google Colab 或官方服务器的配额限制(一天 30 个 jobs),实现 7x24 小时批量预测。
- 自定义参数: 允许调整多序列比对(MSA)深度、模板使用等参数,适配特定科研目标。
本地部署的方法
核心方案:LocalColabFold github.com/YoshitakaMo/localcolabfold
LocalColabFold 是 AlphaFold2 的一个经过优化的本地版本,它将原本复杂的部署过程简化为“一键脚本”,并支持轻量化运行。 为什么它能“轻量化”?
-
云端 MSA 搜索(可选):AlphaFold 最占空间的是几 TB 的基因序列数据库。LocalColabFold 默认可以调用 ColabFold 的公共服务器进行序列比对(MSA),你本地不需要下载那 2.5 TB 的数据。
-
MMseqs2 优化:如果你选择本地搜索,它使用比原版更快的 MMseqs2 算法,内存占用更低,速度提升数十倍。
-
显存优化:支持在消费级显卡(如 RTX 3060/4060 8GB/12GB)上运行中短序列。
步骤如下(假设在 Windows 环境下部署):
- 安装 Windows Subsystem for Linux
wsl.exe --install ubuntu- 启动 WSL 后更换国内镜像
echo '# 默认注释了源码镜像以提高 apt update 速度,如有需要可自行取消注释deb https://mirror.sjtu.edu.cn/ubuntu/ noble main restricted universe multiverse# deb-src https://mirror.sjtu.edu.cn/ubuntu/ noble main restricted universe multiversedeb https://mirror.sjtu.edu.cn/ubuntu/ noble-updates main restricted universe multiverse# deb-src https://mirror.sjtu.edu.cn/ubuntu/ noble-updates main restricted universe multiversedeb https://mirror.sjtu.edu.cn/ubuntu/ noble-backports main restricted universe multiverse# deb-src https://mirror.sjtu.edu.cn/ubuntu/ noble-backports main restricted universe multiverse
# 以下安全更新软件源包含了官方源与镜像站配置,如有需要可自行修改注释切换# deb https://mirror.sjtu.edu.cn/ubuntu/ noble-security main restricted universe multiverse# # deb-src https://mirror.sjtu.edu.cn/ubuntu/ noble-security main restricted universe multiverse
deb http://security.ubuntu.com/ubuntu/ noble-security main restricted universe multiverse# deb-src http://security.ubuntu.com/ubuntu/ noble-security main restricted universe multiverse
# 预发布软件源,不建议启用# deb https://mirror.sjtu.edu.cn/ubuntu/ noble-proposed main restricted universe multiverse# # deb-src https://mirror.sjtu.edu.cn/ubuntu/ noble-proposed main restricted universe multiverse' | sudo tee /etc/apt/sources.list #更换 SJTU 软件源镜像- 安装 CUDA 等前置软件
sudo apt install -y curl wget git cuda-toolkitnvcc --version # 检查 CUDA 是否安装成功- 使用安装脚本安装 LocalColabFold
wget https://raw.githubusercontent.com/YoshitakaMo/localcolabfold/main/install_colabbatch_linux.shbash install_colabbatch_linux.sh编辑 ~/.bashrc
export PATH="~/colabfold-conda/bin:$PATH" # 设置环境变量# 设置 TensorFlow 环境变量,以防超显存export TF_FORCE_UNIFIED_MEMORY="1"export XLA_PYTHON_CLIENT_MEM_FRACTION="4.0"export XLA_PYTHON_CLIENT_ALLOCATOR="platform"export TF_FORCE_GPU_ALLOW_GROWTH="true"这样 LocalColabFold 就安装完成了。
使用方法
colabfold_batch input outputdir/ # input 和 outputdir 分别是存放输入和输出的目录(input 也可以是文件)# 输入文件支持 fasta,csv,a3m 格式其他的运行 Flag 参见项目文档 README
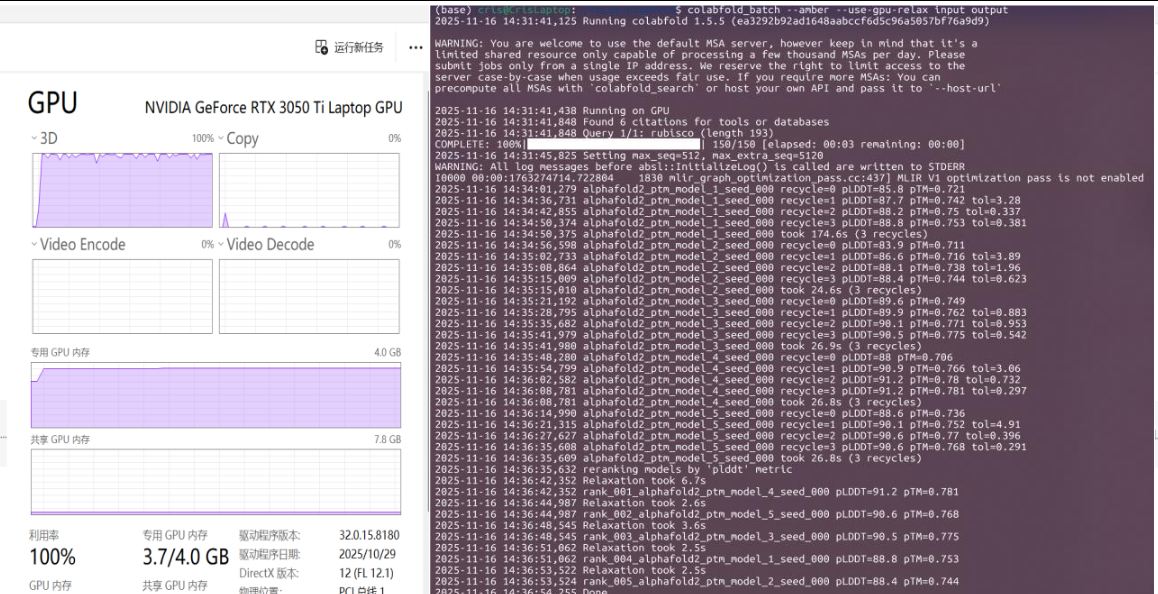
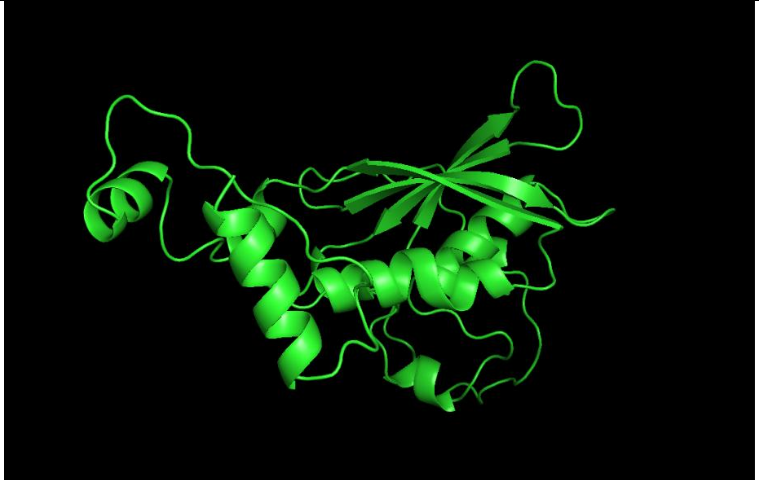
得到的蛋白质结构使用 PyMOL 可视化:
还有没有其他部署方式?
除了使用 LocalColabFold,也可以直接使用 Docker 部署 ColabFold。 可以参考链接:https://github.com/sokrypton/ColabFold/wiki/Running-ColabFold-in-Docker
Docker 部署更为便捷,但是由于国内网络环境限制,Docker 无法从官网 Pull 镜像,故不采纳。
使用 LocalColabFold 本地部署 AlphaFold 2
https://www.cristime.top/posts/localcolabfold/ 